e-DNA – eeee?
 Mój przedostatni wpis był poniekąd sprawozdaniem z warsztatów monitoringowych, w których brałem udział, i tak się składa, że ten też taki będzie. Tym razem byłem na warsztatach dotyczących perspektyw technik identyfikacji molekularnej organizmów w ocenie stanu środowiska wodnego.
Mój przedostatni wpis był poniekąd sprawozdaniem z warsztatów monitoringowych, w których brałem udział, i tak się składa, że ten też taki będzie. Tym razem byłem na warsztatach dotyczących perspektyw technik identyfikacji molekularnej organizmów w ocenie stanu środowiska wodnego.
Warsztaty odbyły się na początku maja we Wiedniu na Uniwersytecie Przyrodniczym (BOKU) i były zorganizowane dla naukowców, którzy często nie najlepiej rozumieją uwarunkowania państwowego monitoringu środowiska, i dla ekspertów od monitoringu, którzy niekoniecznie znają najnowsze osiągnięcia ekologii, zwłaszcza ekologii molekularnej. (Nieprzypadkowo napisałem „we Wiedniu”. Wiem, że w niektórych dialektach polskiego, łącznie z tym, który w ostatnich stuleciach jest podstawą wersji urzędowej i literackiej, przeważa wersja „w Wiedniu”, ale dla użytkowników mojego dialektu stolica przez sto parędziesiąt lat była właśnie we Wiedniu, więc wiem lepiej, jak mam to mówić i pisać).
Fakt, że gatunki czy większe taksony można rozpoznawać metodami molekularnymi, czy to przy użyciu swoistych dla gatunku odcinków DNA, czy białek (np. izozymów), nie zaskakuje. Już w czasie studiów miałem z tym do czynienia i już wtedy zacierał się podział na kaloszowców i fartuchowców, o którym kiedyś pisałem. Współczesne seriale kryminalne w ogóle zaś dają wrażenie, że wystarczy wrzucić trochę materiału biologicznego do probówki i po paru godzinach ma się kompletny portret dawcy tego materiału.
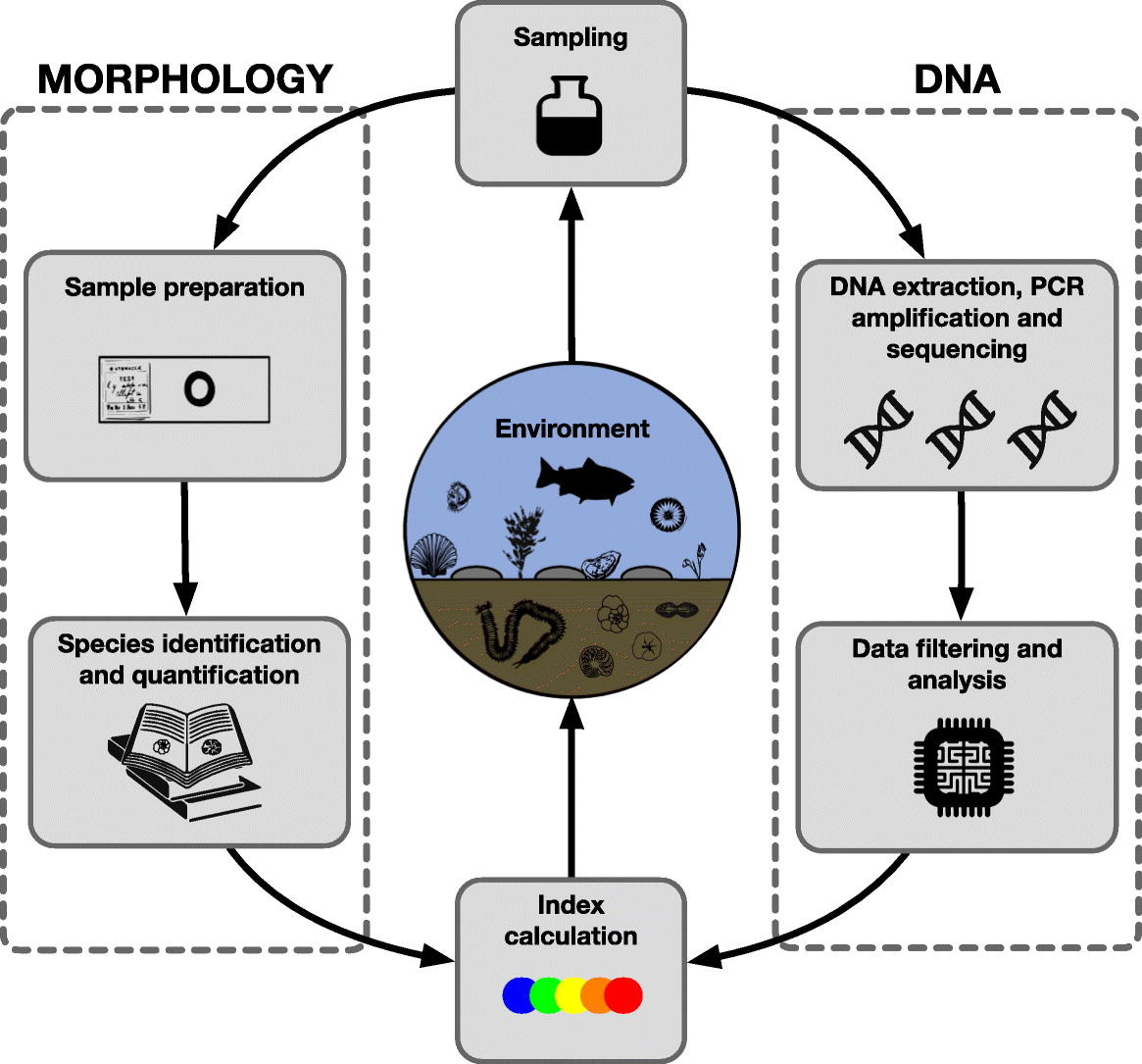
Praktyka jest w wielu aspektach dużo bardziej skomplikowana i jeszcze niedopracowana, ale w innych jest nawet bardziej zaawansowana, niż się może wydawać. Dotyczy to badania metagenomów, w tym środowiskowego DNA (e-DNA). Pobiera się np. biofilm (czytelnicy mogą go znać jako zielony nalot na ścianach akwariów) i po dość rutynowej obróbce w laboratorium biologii molekularnej można dostać może nie portret, ale spis gatunków glonów, które ten nalot tworzą.
Tradycyjnie taki spis dostałoby się po żmudnej obróbce w laboratorium hydrobiologicznym, której pierwszy etap polegałby na sedymentowaniu, odwirowywaniu itd. glonów, oddzielaniu ich od materiału, takiego jak piasek czy muł. Dzieleniu próby na dwie grupy – jedną do analizy okrzemek, którą się przez parę(naście) godzin praży w silnym utleniaczu (np. perhydrolu lub kwasie siarkowym), drugą do analizy pozostałych glonów. Następnie robieniu z tych prób preparatów mikroskopowych i ich dokładnemu oglądaniu.
W trakcie tego oglądania w mikroskopie (żeby to miało sens: to nie jest mikroskop szkolny, tylko taki kosztujący około 100 tys. zł) próbuje się układ prążków, płytek ściany komórkowej czy innych elementów anatomicznych dopasować do obrazków lub zdjęć w kluczach do oznaczania gatunków. Gdy się uda, zapisuje się wystąpienie osobnika danego gatunku i przechodzi do kolejnego osobnika.
Pierwsze wymaga prócz sprzętu i odczynników sumiennego technika laboratoryjnego obsługującego ten sprzęt. To drugie – przede wszystkim eksperta z wprawnym okiem, choć oczywiście obróbka laboratoryjna też ma znaczenie. To pierwsze wydaje się więc prostsze i w miarę tanienia sprzętu i odczynników pewnie będzie zdobywać coraz większą popularność. W badaniach naukowych już zdobywa, w badaniach monitoringowych – jeszcze nie bardzo. Oprócz dość oczywistego czynnika, jakim jest ostrożność przed wprowadzaniem nowości (a więc czegoś nierutynowego, o nie do końca potwierdzonej jakości wyników) do badań, pod którymi podpisuje się urzędnik, są inne, całkiem merytoryczne powody.
Spis gatunków jest ciekawą informacją. Nie tylko katalogową. Dzięki niemu możemy prześledzić inwazje gatunków czy ich lokalne wymieranie. Jakiś gatunek w danym miejscu i czasie występuje albo nie. Jako wskaźnik jakości środowiska jest to jednak informacja ułomna. Owszem, jeżeli mamy z jednego jeziora spis gatunków, w którym są same gatunki wyjątkowo wrażliwe na zanieczyszczenia, a z drugiego takie, w którym są same gatunki znoszące wszystko – mamy niezłą informację. To jednak sytuacja wyjątkowa dla warunków referencyjnych lub kompletnie zdegradowanych.
Zwykle jest sytuacja pośrednia – gdy na spis trafią i gatunki wskazujące na wodę czystą, i na zanieczyszczoną. Jeżeli więc mamy spis, w którym znajdzie się rak szlachetny i gnojka wytrwała (przeskoczyłem z glonów na bezkręgowce) albo sielawa i karaś, niewiele wiemy. Ale gdy wiemy, że w jednej próbie mamy sto raków i jedną gnojkę, a w drugiej jednego raka i sto gnojek, możemy powiedzieć coś więcej. Podobnie gdy mamy próbę jednogatunkową, dajmy na to nitkowatej sinicy, której w jednym poborze złowiliśmy np. jedną nić, a w drugiej tysiąc nici – też możemy oszacować żyzność (a więc prawdopodobne zanieczyszczenie) wody. Liczebność ma znaczenie. Liczebność względna (między gatunkami) i bezwzględna.
O ile z wykrywaniem gatunków metody molekularne dają sobie radę lepiej lub gorzej, o tyle z szacowaniem liczebności organizmów dają sobie radę tylko gorzej. Intuicyjnie można przypuszczać, że im więcej osobników danego gatunku, tym więcej kopii DNA znajdzie się w próbie i tym więcej odczytów markerów molekularnych. Trochę tak, ale tylko trochę. Rzeczywiście, jest to jakoś proporcjonalne, ale na pewno nie jest to banalna zależność. Liczebności absolutnej nie da się w ten sposób na razie obliczyć. Od biedy po pewnych przeliczeniach przy różnych założeniach można próbować szacować liczebność względną gatunków. Te przeliczenia mają np. uwzględnić to, że w świecie glonów nawet jednokomórkowych z tej samej grupy różnice w rozmiarze osobników mogą być wielokrotne, a więc większa liczba odczytów markerowej sekwencji DNA nie musi oznaczać większej liczebności osobników.
Całkiem obiecujące są próby szacowania względnej liczebności dużych organizmów wodnych, takich jak ryby. Jeżeli pobierzemy próby z dziesięciu punktów jeziora i znajdziemy DNA płoci w sześciu z nich, szczupaka w dwóch, a leszcza w dziewięciu, uznanie, że mniej więcej takie są proporcje w liczebności ryb w tym jeziorze, jest nie tak dalekie od prawdy. Przynajmniej nie tak dalekie, żeby nie dało podobnych rezultatów w ocenie stanu ichtiofauny. Oczywiście tu też trzeba pewnych założeń, inaczej trzeba traktować odczyt gatunków samotniczych, a inaczej tworzących duże ławice. Prawda jest taka, że naprawdę samotnicze, pojedyncze osobniki i tak nie zostaną wykryte.
Niemniej z różnych badań wynika, że o ile w tradycyjnych badaniach składu ichtiofauny zwykle w pojedynczym odłowie znajduje się – powiedzmy – połowę gatunków w danym akwenie występujących i w miarę kompletny spis można utworzyć dopiero po paru latach badań, o tyle badanie markerów DNA pozwala na wykrycie wszystkich tych gatunków w jednej serii poborów.
Ale zaraz – po co identyfikować ryby po markerach DNA? Nie trzeba wielkiego specjalisty, że rozpoznać te kilka, góra kilkadziesiąt gatunków ryb, których się można spodziewać w danym jeziorze czy rzece. Rzecz w tym, że do wykorzystania markerów molekularnych nie trzeba mieć prób biologicznych pobranych wprost z organizmów. Wystarczy tzw. e-DNA, czyli DNA pływające swobodnie w wodzie. W filmach detektywistycznych trzeba mieć a to cebulkę włosa, a to wyściółkę jamy ustnej. Jeżeli chce się zidentyfikować osobnika, to tak. Żeby stwierdzić gatunek, wystarczy mieć trochę komórek z naskórka, które spontanicznie odpadają. Kręgowce tracą naskórek w każdym momencie. Niektóre kręgowce i bezkręgowce wytwarzają taki czy inny śluz. W wodzie pływa ostatecznie tyle DNA, że wystarczy parę litrów (z rozwojem techniki coraz mniej), żeby zrobić analizę i dostać spis gatunków.
DNA poza żywym organizmem może przetrwać w wystarczającej formie tydzień, dwa. W wiecznej zmarzlinie może nawet trwać tysiące lat. W osadach w warunkach chłodu, braku światła i braku utleniaczy może też trwać długo i są już robione analizy składu gatunkowego jezior na przestrzeni stuleci. W rzece może przenieść się kilka kilometrów, ale zwykle w wystarczającej do analiz ilości i formie to co najwyżej kilkaset metrów, więc ryzyko wykrycia gatunku, który żyje w nieco innym miejscu (np. w potoku wpadającym do badanej rzeki), jest niewielkie. Stąd też opisana wcześniej możliwość szacowania względnej liczebności na podstawie liczby miejsc ze stwierdzeniem. W istocie DNA wcale nie jest unoszone równomiernie po akwenie i gdy w jeziorze są np. plosa bardziej eutroficzne i oligotroficzne, da się to poznać po znajdowanym e-DNA ryb.
Z drugiej strony fałszywe wyniki dodatnie wynikające z takiego zanieczyszczenia próby zdarzają się. Bywa, że w jeziorze znajduje się DNA pstrąga tęczowego, powszechnie znajduje się DNA łososia, a nieraz tuńczyka. Nie oznacza to, że te gatunki naprawdę w tym jeziorze żyją. Oznacza to, że fragmenty DNA mogą spokojnie przepasażować przez przewód pokarmowy człowieka (tak naprawdę ludzkie DNA jest jednym z najczęściej identyfikowanych w wodzie), kota, psa czy mewy. Stąd należy do takich stwierdzeń podchodzić z odpowiednią ostrożnością.
Z tym że najczęściej stwierdza się ludzkie DNA – to nie zawsze prawda. Ono mogłoby być wykrywane, ale zwykle nie jest, bo się go nie szuka. I tu jest jedna z wad metod identyfikacji po mikromacierzach DNA. Są metody, które mogą pokazać wszystko, przez co pokazują w praktyce szum informacyjny. W praktyce więc zwykle stosuje się wstępną selekcję i szuka tylko już rzeczy spodziewanych i znanych.
Ponadto nawet gdy nie robi się tego typu selekcji, może ona zachodzić na wstępnym etapie obróbki i jest artefaktem technicznym. Otóż identyfikacja po DNA nie odbywa się na zasadzie poszukiwania igły w stogu siana. Pojedyncza nić DNA z markerową sekwencją ma znikomą szansę być odkrytą. Najpierw DNA się namnaża w procesie PCR. (Wydaje mi się to taką oczywistością, że nie będę tego rozwijał). Nie namnaża się go jednak ot tak, ale wybiera się te fragmenty nici, które zawierają interesującą nas sekwencję. Do tych fragmentów przyczepiane są startery. Fragment bez startera nie będzie replikowany (ewentualnie będzie w nieistotnym stopniu).
I tu też jest pies pogrzebany. Startery są specyficzne dla szukanych sekwencji, a więc w tym przypadku dla markerów gatunków. Jeżeli do probówki wrzucimy tylko startery specyficzne dla szczupaka, nawet jeżeli w próbce w większości będzie DNA płoci, ale będą jakieś ilość DNA szczupaka, ostatecznie dostaniemy dużą ilość DNA szczupaka, a ilość DNA płoci może okazać się niewykrywalna.
Z każdym badaniem biblioteka markerów (i odpowiadających im starterów) może powiększyć się o kolejny gatunek. W przypadku ryb stosowanych w monitoringu w krajach europejskich biblioteka tzw. kodów kreskowych DNA obejmuje już 100 proc. Jednak w przypadku okrzemek fitobentosowych to dopiero 30 proc. Specyficzna sytuacja jest w przypadku bezkręgowców bentosowych, bo znane są markery kilkudziesięciu procent gatunków, przy czym to średnia i w jednych grupach to może zbliżać się do 100 proc., a w innych osiągać ledwie kilkanaście.
Natomiast w wielu krajach obecnie w monitoringu oznacza się bezkręgowce tylko do poziomu rodziny, nie zważając na konkretny gatunek, a markery dla rodzin są już znane w 100 proc. Z niektórymi grupami jest większy problem – mimo wszystko. W zasadzie nie wiem też, jak to jest z dostępnością e-DNA tych grup, u których liczba komórek ciała jest w zasadzie stała i nie ma ciągłego złuszczania się naskórka czy wydzielania śluzu (tak jest w przypadku organizmów zwanych kiedyś obleńcami).
Jak pisałem przy okazji odkrycia w trakcie monitoringu okrzemki Diatoma polonica, czasem występowanie gatunku dotąd nieznanego nie ma większego znaczenia dla klasyfikacji jakości wód, a czasami to właśnie ten nieznany dominuje i decyduje o ocenie. Jeżeli w próbie większość DNA nie da się przypisać do sekwencji zgromadzonych w bazie danych, to może być dla naukowców impuls do dalszych badań, ale dla administracji to problem.
Naukowcy badający DNA (wszystko jedno, czy z pobranych organizmów, czy e-DNA) nieraz tworzą quasi-taksony dla nieznanych sekwencji. W takich badaniach zresztą raczej się mówi o operacyjnych jednostkach taksonomicznych (OTU), a nie o gatunkach. Badacze eksplorujący ten teren otwarcie mówią, że opcja, którą najchętniej przyjęliby urzędnicy, czyli stosujemy identyfikację molekularną, bo jest lepsza niż mikroskopowa, ale tak oznaczone gatunki nadal podstawiamy do już zatwierdzonych w rozporządzeniach wzorów – może nie być najlepsza. Niektórzy uważają, że nieważne, czy dana sekwencja DNA należy do bździągwy malutkiej czy bździągwielca pospolitego. Ważne, że taką znajdujemy w wodach o średniej zawartości azotu, a drugą w wodach o wysokiej i trzeba stworzyć nowe wskaźniki oparte na mniej lub bardziej enigmatycznych OTU.
Jest jeszcze jeden problem nie do przeskoczenia (przynajmniej na obecnym etapie rozwoju technicznego). W przypadku ryb ważny jest nie tylko zestaw gatunków i ich proporcje. Ważna jest też demografia. Co z tego, że w rzece mamy mnóstwo łososi, a więc gatunku raczej czystolubnego, skoro mamy tylko dorosłe osobniki? Jeżeli nie ma młodych, coś jest nie tak (np. brak miejsc na tarło). W ramowej dyrektywie wodnej jest wpisane wprost, że dobry stan ekologiczny wyraża się też odpowiednią strukturą wiekową ryb. Niektóre kraje w swoich metodach to tak naprawdę ignorują, wiele, w tym Polska, sprowadza to do zasady – osobnik poniżej 15 cm jest młody, a powyżej – dorosły (wiek ryby poznaje się po oczach – im dalej oczy są od ogona, tym ryba starsza). Markery DNA nie rozpoznają wieku, nawet szacowanego w tak uproszczony sposób.
Nikt jednak nie mówi, że metody identyfikacji molekularnej muszą wyczerpywać temat. W przypadku fitoplanktonu jego obfitość bezwzględna jest kluczowa. W wodach morskich jest nawet ważniejsza od składu taksonomicznego. Obecnie mierzy się ją na dwa sposoby – zliczaniem osobników i mnożeniem przez standardową objętość komórki danego gatunku oraz pomiarem stężenia chlorofilu w wodzie. W tym przypadku można by nadal szacować biomasę przez pomiar ilości chlorofilu, a oznaczanie składu taksonomicznego pod mikroskopem zastąpić metodami molekularnymi. W przypadku fitobentosu, bezkręgowców i ryb wystarczy liczebność względna. U tych ostatnich można by wykorzystać metody hydroakustyczne. Obecne echosondy są w stanie oszacować nawet niektóre gatunki, ale to mało wiarygodne, natomiast całkiem nieźle nadają się do szacowania rozmiarów ryb, a to już nie jest dalekie od obecnego uproszczonego szacowania wieku. Tu też byłoby więc pole do jakichś metod hybrydowych.
Niektórzy eksperci są bardzo sceptyczni, zwłaszcza w przypadku ryb. Uważają, że obecne metody, choć pracochłonne, dają lepszą informację. Złowiwszy rybę, można ją wziąć do ręki, zmierzyć (tak jak na zdjęciu w niegdysiejszym wpisie), zważyć, zobaczyć, czy ma jakieś pasożyty, egzemy itd. To znacznie więcej, niż da się dowiedzieć metodami molekularnymi.
Z drugiej strony tradycyjne metody są kontrowersyjne ze względów etycznych i ekologicznych. Ryby po elektropołowie zwykle mimo chwilowego szoku przeżywają, ale nieraz taka obróbka oznacza ich śmierć po paru dniach na skutek otarć skóry itp. rzeczy, które ułatwiają infekcje, łącznie z tym, że np. wyciągnięcie ryby z wody o temperaturze bliskiej zera na rękę o temperaturze trzydziestu paru stopni Celsjusza też nie jest bez znaczenia (fragment z dedykacją dla miłośników metody „złap i wypuść”).
Ryby po połowie sieciami skrzelowymi przeżywają tylko w raportach, w rzeczywistości wracają do środowiska jako martwa materia organiczna. A taki połów trzeba wykonywać regularnie. Gdyby nawet zignorować te obiekcje, pozostaje kwestia kompletności takiego połowu. Otóż taki połów zawsze jest mniej lub bardziej wyrywkowy. Gatunki żyjące przy dnie czy mniej wrażliwe na narkozę elektryczną i tak się nie złapią. W ich przypadku tradycyjne metody wcale nie dają lepszej informacji, bo dla tradycyjnych metod te ryby są niewidzialne. Poza tym przy odpowiednich starterach może okazać się, że dużo lepiej da się oszacować stopień zapasożycenia populacji (nawet jeżeli nie zobaczy się konkretnego pasożyta na konkretnej rybie).
Teoretyzowanie to jedno, a praktyka – drugie. Wiadomo, że na konferencjach i w publikacjach prezentuje się to, co się udało, a nie wspomina o tym, co nie. Niemniej w co najmniej paru przypadkach badań pilotażowych testowano klasyfikację po identyfikacji tradycyjnymi metodami i klasyfikację z użyciem DNA i występuje całkiem duża zbieżność. W związku z tym od dwóch lat Anglia, a od tego roku Szkocja przeszły na metody DNA w ocenie stanu fitobentosu okrzemkowego (Walia i Irlandia Północna nie dały się przekonać i uznały, że w ich skali identyfikacja mikroskopowa będzie tańsza). Na razie to awangarda. Reszta Europy nie musi się spieszyć, choć to pewnie kwestia kilku(nastu) lat, zanim pozostałe kraje zaczną się ku temu skłaniać.
A Polska? Na razie nie wiadomo. Warsztaty zorganizowano w ramach sieci naukowej DNAqua-Net. W tej sieci są polscy naukowcy, ale z tego, co wiem, zajmuje ich raczej bioróżnorodność niż monitoring. Stąd na warsztatach ich nie było i wśród autorów publikacji z tego zakresu też niezbyt się pojawiają. Z kolei naukowcy, którzy dotychczas tworzyli metody monitoringu biologicznego, jak dotąd wydają się przywiązani do metod, które już znają.
ilustracja: Pawłowski i in. (2018), licencja CC BY-NC-ND 4.0
- Hering, D., Borja, A., Jones, J.I., Pont, D., Boets, P., Bouchez, A., Bruce, K., Drakare, S., Hänfling, B., Kahlert, M., Leese, F., Meissner, K., Mergen, P., Reyjol, Y., Segurado, P., Vogler, A., Kelly, M., 2018. Implementation options for DNA-based identification into ecological status assessment under the European Water Framework Directive. Water Research 138, 192–205. https://doi.org/10.1016/j.watres.2018.03.003
- Leese, F., Bouchez, A., Abarenkov, K., Altermatt, F., Borja, Á., Bruce, K., Ekrem, T., Čiampor, F., Čiamporová-Zaťovičová, Z., Costa, F.O., Duarte, S., Elbrecht, V., Fontaneto, D., Franc, A., Geiger, M.F., Hering, D., Kahlert, M., Kalamujić Stroil, B., Kelly, M., Keskin, E., Liska, I., Mergen, P., Meissner, K., Pawlowski, J., Penev, L., Reyjol, Y., Rotter, A., Steinke, D., van der Wal, B., Vitecek, S., Zimmermann, J., Weigand, A.M., 2018. Why We Need Sustainable Networks Bridging Countries, Disciplines, Cultures and Generations for Aquatic Biomonitoring 2.0: A Perspective Derived From the DNAqua-Net COST Action, w: Bohan, D.A., Dumbrell, A.J., Woodward, G., Jackson, M. (Eds.), Advances in Ecological Research, Next Generation Biomonitoring: Part 1. Academic Press, pp. 63–99. https://doi.org/10.1016/bs.aecr.2018.01.001
- Pawlowski, J., Kelly-Quinn, M., Altermatt, F., Apothéloz-Perret-Gentil, L., Beja, P., Boggero, A., Borja, A., Bouchez, A., Cordier, T., Domaizon, I., Feio, M.J., Filipe, A.F., Fornaroli, R., Graf, W., Herder, J., van der Hoorn, B., Iwan Jones, J., Sagova-Mareckova, M., Moritz, C., Barquín, J., Piggott, J.J., Pinna, M., Rimet, F., Rinkevich, B., Sousa-Santos, C., Specchia, V., Trobajo, R., Vasselon, V., Vitecek, S., Zimmerman, J., Weigand, A., Leese, F., Kahlert, M., 2018. The future of biotic indices in the ecogenomic era: Integrating (e)DNA metabarcoding in biological assessment of aquatic ecosystems. Science of The Total Environment 637–638, 1295–1310. https://doi.org/10.1016/j.scitotenv.2018.05.002
Komentarze
(1) Co to za przerażająco drogi mikroskop, a ściślej: co on umie? W mikroskopii optycznej moją wyobraźnię przekracza wszystko powyżej Biolara z kontrastem Nomarskiego.
(2) Przy okazji zadam głupie pytanie: wyczytałem dziś nad ranem, że jak w ZSRR pobudowali zapory na Wołdze i zbiorniki w Wołgogradzie i Kujbyszewie, zatamowało to migracje ryb, gdyż „ryby te nie chciały korzystać ze specjalnie zainstalowanych wind” (Zbigniew Wójcik, Katastrofy zapór wodnych, Warszawa 1966, s. 61).
Czy to prawda? Windy dla ryb? Chodzi o jakieś śluzy?
Załączniki do SIWZ pokazują, co taki mikroskop ma umieć (ma np. umieć filmować) http://www.gios.gov.pl/bip/zamowienie_publiczne.php?id=471
Windy dla ryb: https://www.youtube.com/watch?v=W4vC6u_tJXc
https://www.youtube.com/watch?v=bpMlkbk1p30
https://www.youtube.com/watch?v=jbWWRsmRLL8
Kontynuując temat głupich pytań i wątpliwości:
1) Po co się utlenia te okrzemki?
2) Naskórek, keratynizując się, traci w większości organella, w tym jądro z DNA.
3) Czy nie było jakiejś metody oceny ilościowej namnażanego DNA? COś w stylu Real Time PCR?
@ppanek
Pięknie dziękuję. Widzę, że techniki mikroskopowe nie zmieniły się radykalnie. A windy – nie sądziłem, że to są po prostu i naprawdę windy.
1) Żeby pozbyć się materii organicznej, a przez to wyeksponować prążkowanie krzemionkowego pancerzyka
2) Od ciała odpada w wodzie najwyraźniej nie tylko ten najbardziej skeratynizowany naskórek. Co tam dokładnie zaś odpada od ryb, które mają w większości kostne (a więc skórne, a nie naskórkowe) łuski, oraz od bezkręgowców to tak do końca sam nie wiem. Cześć e-DNA unosi się też ze zwłok. Wtedy pozostają do dyspozycji raczej krótkie odcinki.
3) Najwyraźniej to tak nie działa. Gdyby działało, byłby to przełom w monitoringu.
Myślę, że w mechanizmie wind najbardziej frapujące może być to, że w odróżnieniu od innych przepławek, gdzie ryby przepływają, kiedy chcą, tu muszą się najpierw zebrać w jednym miejscu i czasie. Właściwie, to sam nie wiem, jak się to do końca dzieje. Jak w każdej innej porządnej przepławce tu pewnie przede wszystkim się wabi do takiej windy (po pierwsze stawiając barierę na drodze do turbin, np. kurtynę elektryczną, po drugie – prądem, w sensie nurtem – wabiącym – ryby wędrowne podążają instynktownie pod prąd, więc gdy stworzy się w korycie rzeki/kanału odpowiedni strumień, podążają nim.)
Faktem jest, że gdy na studiach próbowałem robić PCR, to mi to ogólnie nie działało…
Okrzemkowe pancerzyki są bardzo, bardzo piękne.
Ciekawe, dlaczego radzieckie ryby (z kontekstu wynikało, że windy zainstalowano z myślą o jesiotrach) nie chciały korzystać z radzieckich wind.
Widocznie to sabotażyści byli, ci jesiotrzy (a może i drugiej świeżości). Towarzysze inżynierowie zaprojektowali windy (w wielu domach nie ma, a w zaporze była), a ryby nie chciały korzystać. Opcji, że towarzysze inżynierowie nie zastosowali prądu wabiącego o odpowiedniej sile, ciężko dać wiarę.
Cytat:
„Podczas przebudowy Wrocławskiego Węzła Wodnego w 2016 r. zbudowano szereg przepławek dla ryb wędrownych na rzece Odrze. Odrze. Trzy przepławki wyposażono w instalacje monitorujące przemieszczanie się ryb.(…) Podczas odbioru technicznego konstrukcji w styczniu 2017 r. dokonano próby z rybami, aby sprawdzić skuteczność działania instalacji monitorującej. (…) Instalacja monitorująca zarejestrowała ok. 60 % przemieszczających się ryb. Trafność przypisania egzemplarza do gatunku była mała (program określił ryby jako brzanę, pstrąga, łososia). Kierunek przemieszczania się ryb był określany prawidłowo. (…) Ze względu na trwające w czasie badań prace nad dostosowaniem prędkości nurtu wody w przepławce do wymagań większości gatunków ryb odrzańskich, nie udało się zaobserwować ryb przepływających pod prąd wody. Prędkość nurtu w przepławce oceniono na ok. 3 – 4 m/s, co przekracza wielokrotnie możliwości pokonania takiego prądu wody przez krajowe gatunki
ryb.” To nie ZSRR w latach 60. tylko kraj unijny współcześnie. (A ja pisałem niedawno o drożności zapór nie tylko dla ryb, ale i rumowiska)
A pancerzyki okrzemek najlepiej wyglądają pod mikroskopem elektronowym, ale takich w monitoringu się nie stosuje.
@ppanek
Może radzieckie jesiotry należało wabić na opera omnia Lenina, nie wiem.
A w obecnym ustroju, jeśli w przepławce prąd jest za silny, wystarczy wezwać kołcza, który rybie wytłumaczy, że jest za mało nastawiona na sukces.
Prezentacje z warsztatów są dostępne: https://circabc.europa.eu/w/browse/0de16346-3a2e-493d-92c3-5a9852e69b58